稳健且准确,AlphaFold结合两种AI方法,实现蛋白质化学位移自动分配
编辑 | 绿萝
化学位移分配对于基于核磁共振 (NMR) 的蛋白质结构、动力学和相互作用研究至关重要,可提供重要的原子级见解。然而,获得化学位移分配是劳动密集型的并且需要大量的测量时间。
为了解决这一限制,苏黎世联邦理工学院(ETH Zurich)的研究人员之前提出了 ARTINA——一种用于自动分配二维 (2D)–4D NMR 谱的深度学习方法。
近日,研究人员提出了一种将 ARTINA 与 AlphaFold 和 UCBShift 相结合的综合方法,能够减少实验数据、提高准确性并增强大型系统的稳健性,从而实现化学位移分配。
研究证明,与使用所有实验可用的 NMR 数据(平均每个蛋白质 10 个 3D 光谱,91.37%)的纯 ARTINA 运行相比,五个 3D 光谱产生更准确的分配(92.59%),从而大大减少了所需的测量时间。研究还报告了在多达 500 个残基的大型合成系统中提高了分配准确性。
该研究以《Time-optimized protein NMR assignment with an integrative deep learning approach using AlphaFold and chemical shift prediction》为题,于 2023 年 11 月 22 日发布在《Science Advances》上。
核磁共振 (NMR) 光谱是一项关键的分析技术,可提供蛋白质结构、动力学和相互作用的详细信息。
使用本质上存在的核自旋探针可以同时获得大量单个原子位置的这些数据。为了实现这种原子分辨率,必须将核自旋的共振频率(以化学位移表示)归因于蛋白质中的各个原子。这种化学位移分配是大多数蛋白质 NMR 研究的一项关键任务。寻找化学位移分配非常耗时并且需要专业知识。
为了加速 NMR 化学位移分配来改变这种情况,人们应该减少所需光谱的数量,并在不影响结果可靠性的情况下实现分析的自动化。
在此,研究人员提出了一种方法,通过利用机器学习的最新进展并将三维(3D)蛋白质结构中包含的信息有效地纳入分配过程来实现这一目标。后者用于取代必须从额外的核磁共振谱中收集的信息。
通用自动分配方法 FLYA 最近被嵌入到 NMR 应用人工智能方法 (ARTINA) 和 NMRtist 网络服务器中,它们使用机器学习进行可视化光谱分析和其他任务,以自动完成从原始光谱到分配和 3D 结构的蛋白质核磁共振数据分析的整个过程。ARTINA 基本上减少了蛋白质化学位移分配的工作量,减少了样品制备和光谱测量的工作量。
然而,平均而言,需要使用超过 13 个多维 NMR 谱才能获得这些结果,这相当于使用传统采集方案对每个蛋白质进行 2 周以上的 NMR 测量时间。考虑到 ARTINA 算法的计算时间通常小于 2 小时,并且 NMR 波谱仪的操作比计算机的操作成本更高且要求更高,因此减少用于分配的光谱数量是提高生物分子核磁共振项目效率的明显策略。
蛋白质 3D 结构的知识主要可以通过两种方式支持化学位移分配:通过更真实地预测核欧佛豪瑟效应频谱(NOESY)中的预期交叉峰,以及通过基于结构的化学位移值的预测。这变得尤为重要,因为 AlphaFold 现在可以准确预测大多数结构化蛋白质的 3D 结构。
ARTINA 与 AlphaFold 和 UCBShift 相结合的综合方法基于此,研究人员建立了一种基于机器学习的综合方法,用于基于结构的 NMR 化学位移分配。评估了其性能并确定用于分配主链酰胺基团或所有化学位移的最佳光谱集。
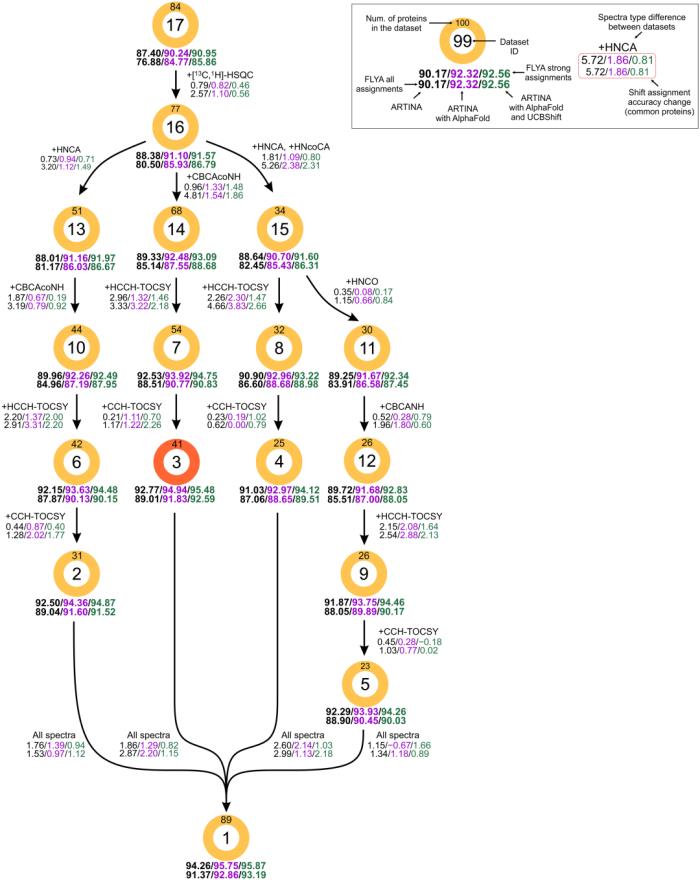
从 ARTINA 原始出版物的 89 种蛋白质的 1170 个实验光谱开始,研究定义了 25 组不同的输入光谱,用于 ARTINA 的自动分配计算。
化学位移分配的准确性在三种不同的情况下进行了评估:通过「经典」三共振谱对主链酰胺基团进行分配,通过 3D NOESY 和三共振谱对主链酰胺基团进行分配,以及完成主链和侧链化学位移的分配,并在每种情况下比较 ARTINA 在没有结构输入的情况下获得的分配结果,或仅使用结构生成预期的 NOESY 交叉峰,或者使用 UCBShift(一种化学位移计算程序)方法将该结构用于预测化学位移。
此外,研究使用仅用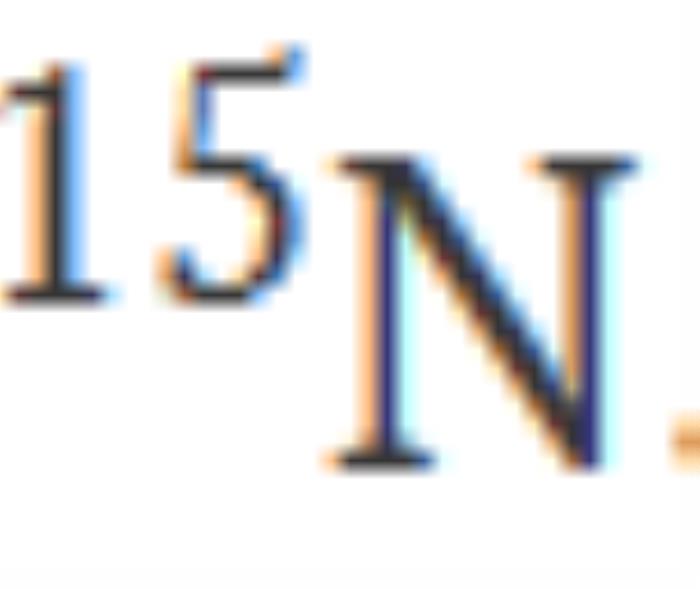 标记样品记录的 NMR 谱测试了主链酰胺分配的可行性,这可以构成昂贵的
标记样品记录的 NMR 谱测试了主链酰胺分配的可行性,这可以构成昂贵的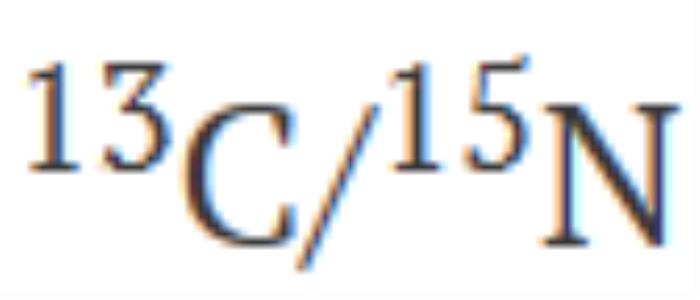 双标记的替代方案。
双标记的替代方案。
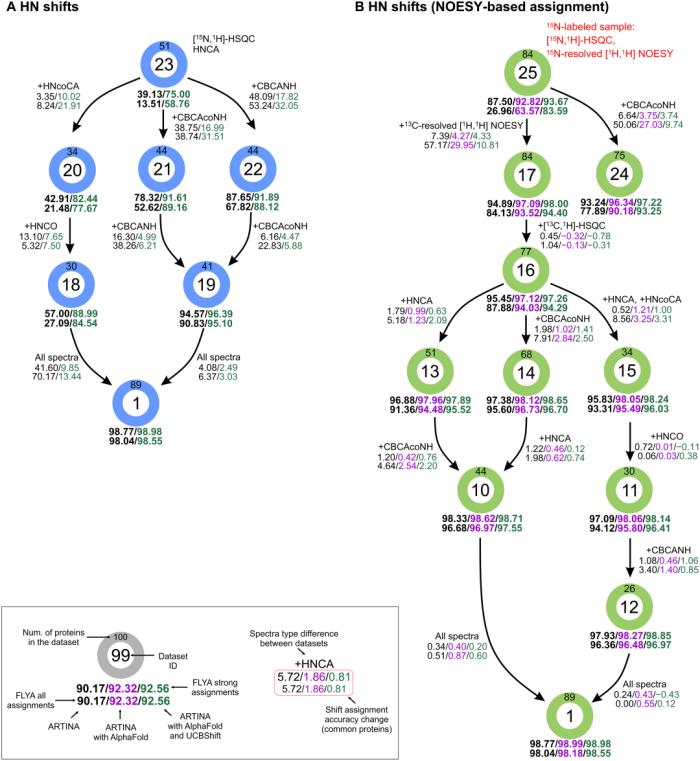
图 2:实验数据量对主链酰胺化学位移分配准确性的影响。(来源:论文)
研究人员使用 3DRobot 生成的大量折叠良好且包装良好的 decoys 来评估输入结构的准确性对分配的影响,这些 decoys 与实验结构的偏差为 0 到 5Å 均方根偏差(RMSD)。
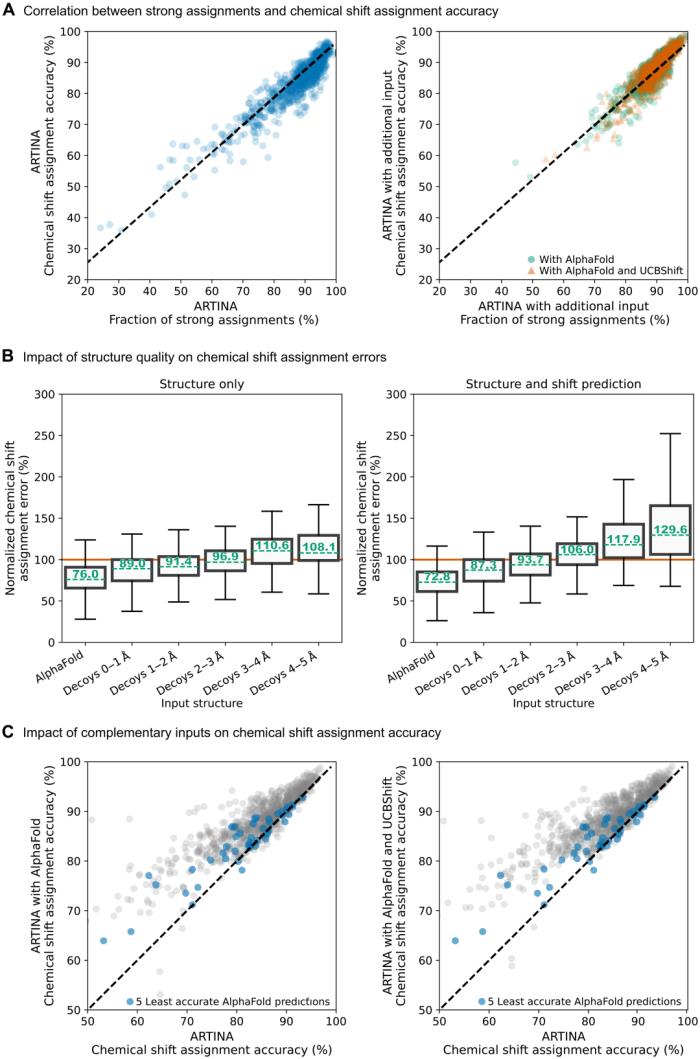
研究人员还评估了综合方法如何分配大型合成生成的蛋白质系统(最多 500 个残基),这些系统目前很少存放在生物磁共振数据库 (BMRB) 数据库中。
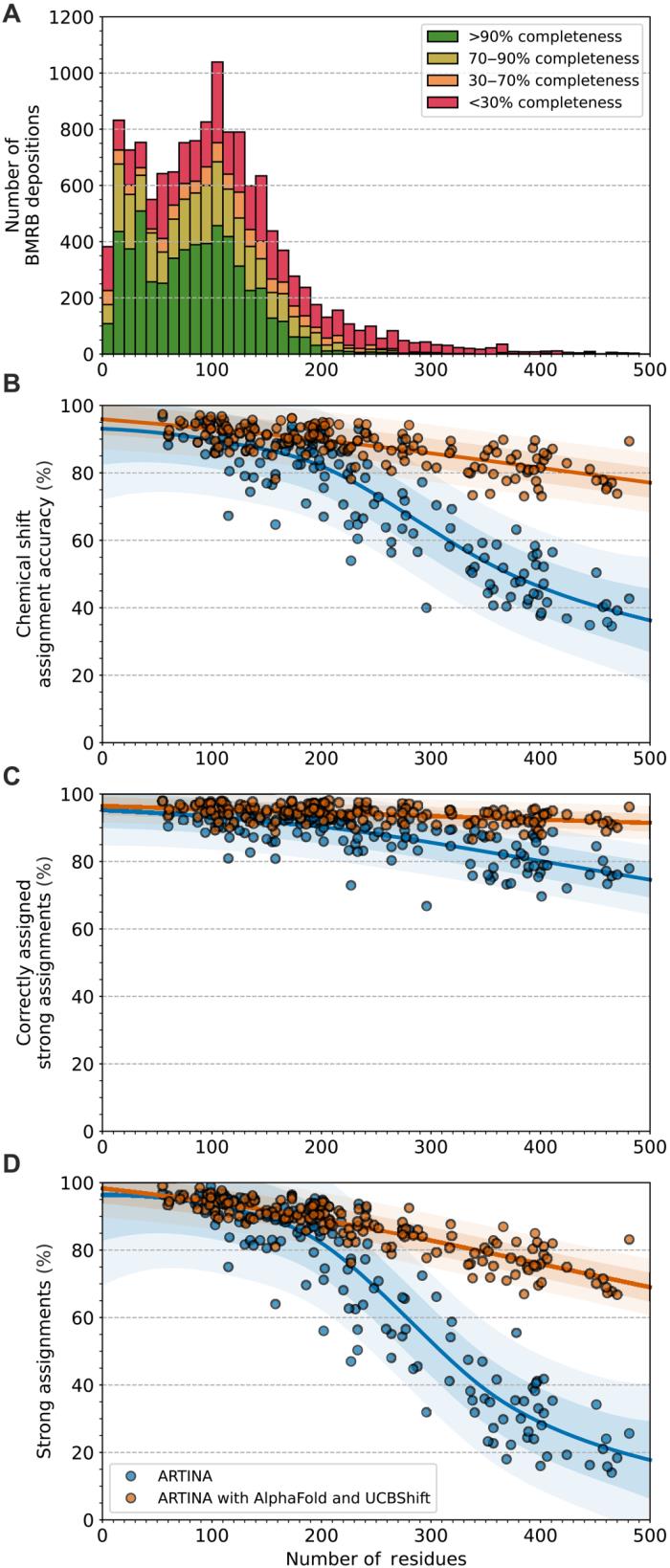
总之,结合该综合方法及其评估,研究人员提出了一套数据驱动的实用建议,用于执行蛋白质的化学位移分配。
综合方法还具有研究其他类型系统的潜力,例如蛋白质复合物、固有无序蛋白质、膜蛋白以及细胞内和固态核磁共振,对于这些系统,机器学习方法与单体系统同样有前景。
- 免责声明
- 本文所包含的观点仅代表作者个人看法,不代表新火种的观点。在新火种上获取的所有信息均不应被视为投资建议。新火种对本文可能提及或链接的任何项目不表示认可。 交易和投资涉及高风险,读者在采取与本文内容相关的任何行动之前,请务必进行充分的尽职调查。最终的决策应该基于您自己的独立判断。新火种不对因依赖本文观点而产生的任何金钱损失负任何责任。





 新火种 2023-12-01
新火种 2023-12-01